|
|
Le but de ce travail est de réaliser un outil de conception graphique interactif de réseau de régulation et de reseau métabolique avec une sortie en SBML (langage standard d'échange en bioinformatique) et dans le langage spécifique au simulateur de processus intra-cellulaire HSIM.
Des outils de ce genre existent déjà (notamment CellDesigner) mais sont difficile à adapter aux buts qu'on se propose d'atteindre.
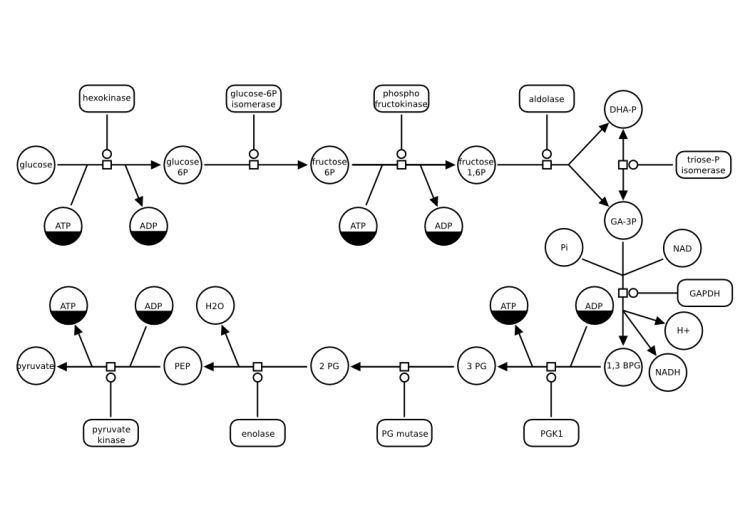
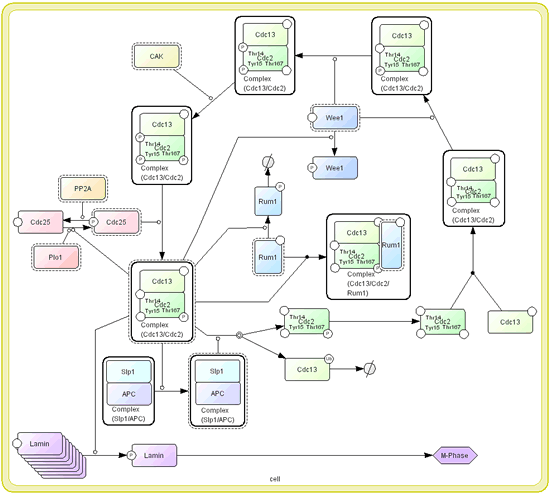
Voila quelques exemples de réseaux qu'on voudrait pouvoir créer interactivement, éditer et sauvegarder :


Pour des raisons de compatibilité avec HSIM, il est demandé de
programmer en C++ pour la partie code et en OpenGL pour le graphique (2D
seulement, 3D si vous avez des idées d'interface 3D!).
Le mieux (mais ce n'est pas obligatoire) serait que vous ayez suivi l'option
de programmation objet avancée.
Un texte plus précis sera sur cette page dans les jours qui viennent, mais vous pouvez me contacter par mail si vous etes interessé.
Patrick AMAR (pa@lri.lri.fr)